 TuttoChimica.it
TuttoChimica.it
Chimica, laboratorio e dintorni...

Duplicazione del DNA
Duplicazione del DNA in eucarioti e procarioti
Dopo la scoperta della struttura del DNA, Watson e Crick ipotizzarono che il doppio filamento del DNA potesse replicarsi utilizzando un meccanismo 'a stampo'. La duplicazione del DNA in due copie uguali, al momento dela divisione cellulare, è fondamentale per consentire il concetto di ereditarietà dei caratteri delle specie. Il modello di duplicazione proposto è detto modello semiconservativo in quanto prevede che ognuno dei due filamenti serva da stampo per il filamento complementare, ottenendo alla fine due DNA identici (modello detto semiconservativo, perchè i due DNA finali avranno entrambi un filamento del 'vecchio' DNA e uno del nuovo).
Diversi sono i 'protagonisti' che intervengono nella duplicazione del DNA:
- proteine elicasi- proteine destabilizzatrici dell'elica
- topoisomerasi
- emielica che funge da stampo
- innesco per la DNA polimerasi
- proteine primasi
- DNA polimerasi
- proteine ligasi
- ATP, GTP, CTP e TTP monomeri
L'elicasi è una proteina in grado di rompere la doppia elica utilizzando energia, sotto forma di una molecola di ATP ogni giro di elica tagliato. Per evitare che, a seguito del taglio, i due filamenti si riappaiano, intervengono le proteine destabilizzatrici dell'elica, che mantengono distaccati i due filamenti. Questo tuttavia comporta una torsione, data la particolare struttura della doppia elica, che aumenta all'avanzare dell'azione dell'elicasi. Intervengono allora degli enzimi, le Topoisomerasi I e II, che tagliano rispettivamente 1 e 2 volte (su un o entrambi i filamenti) che saranno poi da esse ricucitidopo che il filamento di DNA si sarà ruotato su sè stesso in modo da rimuovere le torsioni (superavvolgimenti) che si creano a valle del taglio dell'elicasi.
A questo punto si è formata la cosiddetta forcella di replicazione, ossia un primo tratto di DNA coi due filamenti separati e rilassati (senza superavvolgimenti). Interviene a questo punto il cosiddetto innesco (o primer), un piccolo frammento di filamento di RNA costituito da una decina di nucleotidi che si lega tramite legami a idrogeno al filamento di DNA, in modo da fornire un estremità 3'-OH libera su cui si attaccherà l'enzima DNA polimerasi III. Questo enzima attacca i vari desossiribonucleosidi-trifosfati, in modo che la polimerizzazione avvenga sempre in direzione 5'-->3'. L'enzima assicura la formazione dei legami fosfodiesterici (con perdita del pirofosfato P-P) mano a mano che il filamento nuovo viene ricreato. Poichè l'elicasi svolge il DNA in una direzione sola, un filamento (il filamento 3'-->5') sarà replicato a partire dall'innesco in modo continuativo, invece sull'altro filamento stampo (il 5'-->3') si porteranno varie primasi mano a mano che la forcella di replicazione si apre: da ognuna di queste primasi partirà l'azione delle DNA polimerasi III, che formeranno tanti frammenti di DNA (frammenti di Okazaki) complementari al filamento stampo, sempre in direzione 5'-->3'.
All'avanzare della sintesi dei filamenti complementari avviene anche l'azione delle DNA polimerasi I e II che rimuovono i frammenti di innesco di RNA sostituendoli con analoghi di DNA, e le interruzioni tra i frammenti dei pezzi di DNA sostituiti e il resto del filamento, così come tra i vari frammenti di Okazaki, saranno saldate da un altro enzima detto ligasi.
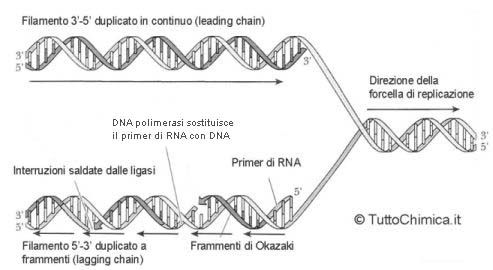
Le DNA polimerasi svolgono quindi una funzione esonucleasica, cioè possono rimuovere, uno alla volta, i nucleotidi dei primer di RNA. Inoltre sono dette correttori di bozze, in quanto possono correggere eventuali errori nell'appaiamento tra le basi dei filamenti nascenti con quelli stampo.
Duplicazione del DNA in eucarioti:
Negli eucarioti il DNA, più lungo e complesso che nei procarioti, è diviso in zone di eucromatina ed eterocromatina. Per favorire il processo, la replicazione parte da svariate forcelle (dette repliconi) poste per tutta la lunghezza del DNA. Queste forcelle non partono tutte contemporaneamente, ma prima compaiono nelle zone di eucromatina e in seguito nelle zone di eterocromatina.
Dobbiamo anche considerare che nel DNA eucariotico troviamo gli avvolgimenti
sugli istoni, e nel DNA finale ogni ottamero sarà costituito da 4 proteine
istoniche vecchie e 4 nuove.
Categorie Articoli e Appunti
• Chimica Generale
•
Chimica Analitica
•
Chimica Inorganica
•
Chimica Organica
•
Biochimica
•
Biologia
•
Genetica